康普森小课堂 | 从全基因组到候选基因还可以这样
GWSS(genome-wide scans of selection,全基因组扫描选择)指通过对自然群体的全基因组进行扫描,寻找并比较具有高度代表性的单倍型信息,分析基因组上存在的特异位点。
GWSS首次应用是在人类基因组进化的研究中[1], 通过比较亚非欧群体间高度纯合的单倍型(extended haplotype homozygosity,EHH),分析各个群体基因组中受到自然选择的基因,这一方法要解决的问题在于区分基因组中的自然选择区段与中性效应。该研究开发了一种新的算法能够分析这种效应,并得出群体中的基因组背离了中性效应,具有明显的自然选择趋势,并利用GO注释信息找到一些可能存在的控制性状变异的候选基因。
GWSS的用途
① 进化分析中寻找基因组上自然选择的足迹
② 补充控制常规性状的基因定位信息(候选基因的选择)
近期案例
近日,在Plant Journal上发表了一篇关于二穗短柄草利用全基因组扫描选择(GWSS)检测位于不同地理位置的群体中的自然选择优势基因[2],并验证了三个抗叶锈病候选基因的存在性,为禾本科植物群体进化及候选基因的选择提供了研究基础。

二穗短柄草(Brachypodium distachyon.)是一种新型的单子叶自花授粉植物,生育期3个月,其基因组虽小到只有272Mb,但与主要粮食作物水稻、小麦、玉米有很好的共线性,且遗传转化技术成熟,是很好的小粒谷物生物学研究的模式植物,其研究模式也是典型禾本科植物的应用研究模型。
研究利用44份自然材料的全基因组测序数据,首先分析了群体结构及遗传多样性,发现这个物种的多个时间和空间尺度上,自然选择正在形成遗传多样性。研究群体可以分为两大类,这两大类群体地理分布具有一致性,分别命名为Western与Eastern群体,它们的多态性及LD衰减值都具有显著差异。
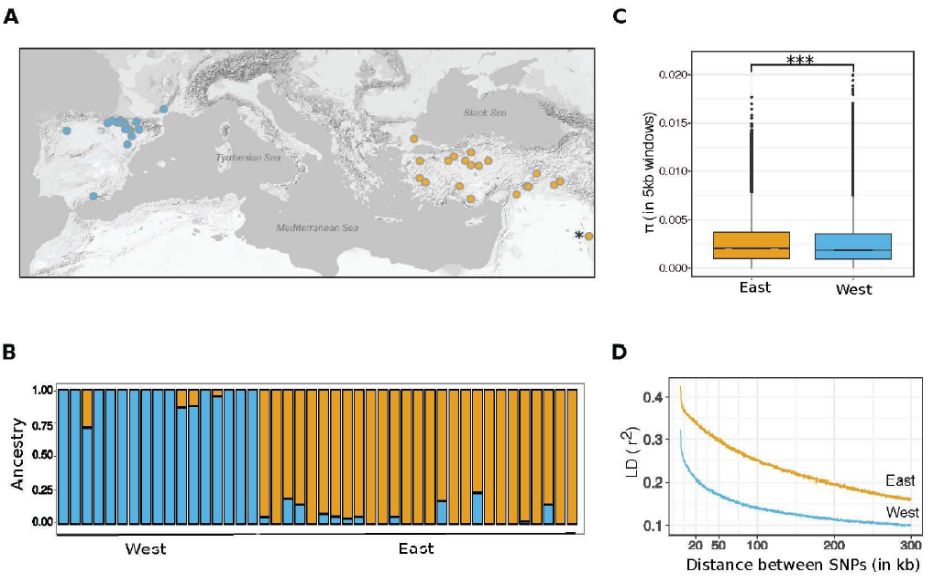
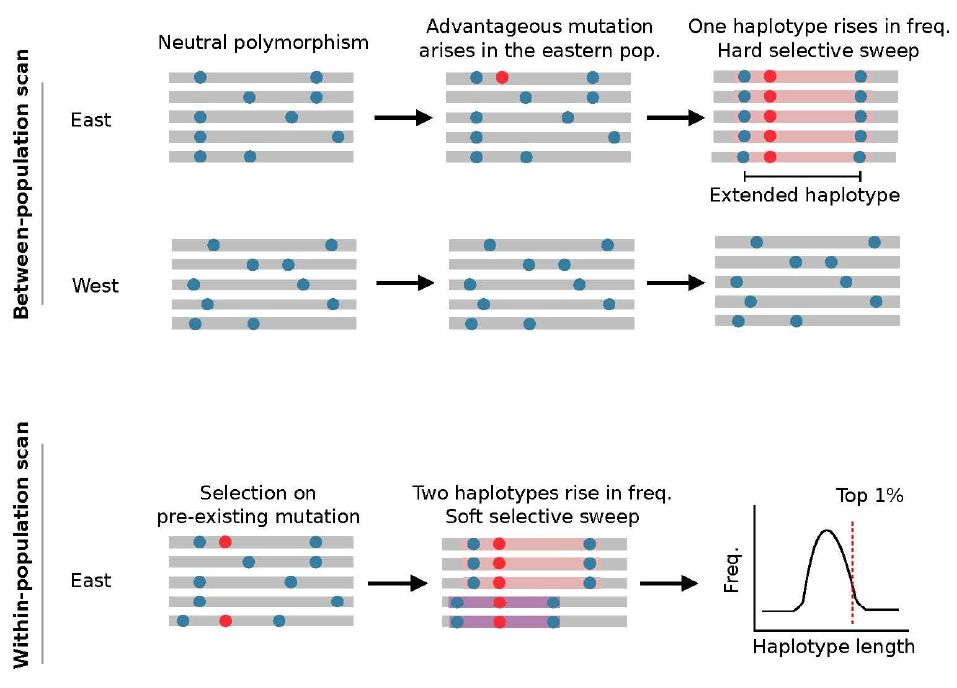
下图是研究人员利用GO注释信息寻找基因组上的over-represented区域,并利用Rsb test[1]进行群体间Hard Scan分析,利用H-Scan[3]进行群体内Soft Scan分析,结合diploS/HIC方法筛选基因组上自然选择的优势基因,利用H-scan 和 Rsb tests检测,在Western群中分别找到了164与312个基因,在Eastern群体中分别找到了258与644个基因。
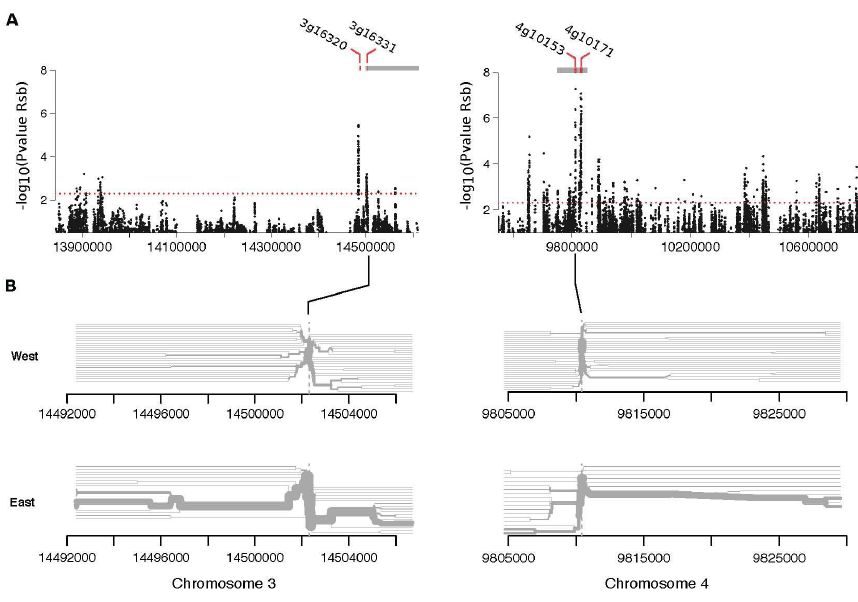
通过结合前人的抗叶锈病QTL定位数据[4],重点分析了选择到的抗病候选基因。这些具有抗病特征的基因正好位于QTL区间内,验证了该位点内抗病候选基因的存在性,为后期的功能验证奠定遗传基础。
GWSS方法主要基于完整的全基因组序列及未经人工驯化的自然群体,之前在人类及动物上研究比较多,此次在二穗短柄草上的研究开启了禾本科全基因组扫描选择研究的大门,随着测序技术越来越成熟,相信GWSS的应用会越来越多。
参考文献
[1] K. Tang, K. R. Thornton, M. Stoneking. A new approach for using genome scansto detect recent positive selection in the human genome. Plos Biology.2007.
[2] Y.Bourgeois, C. Stritt, J.C. Walser, et al. Genome-wide scans of selection highlight the impact of biotic and abiotic constraints in. Plant Journal. 2018.
[3] F. Schlamp, J. Made., R. Stambler, et al. Evaluating the performance of selection scans to detect selective sweeps in domestic dogs.Mol Ecol. 2016
[4] Barbieri M, Marcel TC, Niks RE, et al. QTLs for resistance to the false brome rust Puccinia brachypodii in the model grass. Genome. 2012.

 PREV
PREV
