【Nature子刊】小麦高密度遗传图谱构建及穗粒数QTL定位

客户信息:原中国科学院遗传与发育生物学研究所农业资源研究中心的崔法博士,现就职于鲁东大学农学院 影响因子:4.259 康普森生物提供:小麦660K SNP基因分型芯片检测技术 660K芯片构建小麦高密度遗传图谱填补目前空白 原中国科学院遗传与发育生物学研究所农业资源研究中心的崔法博士(现就职于鲁东大学农学院)采用小麦660K SNP基因分型芯片技术构建了基于高密度SNP的遗传图谱。并对穗粒数进行了QTL定位。相关研究成果“Utilization of a Wheat660K SNP array-derived high-density genetic map for high-resolution mapping of a major QTL for kernel number”发表在Nature子刊《Scientific Reports》上,影响因子4.259(该研究中小麦660K SNP基因分型芯片检测由北京康普森生物公司提供)。
高密度遗传连锁图谱是作物性状形成分子机制研究的基础。小麦基因组大而复杂阻碍了高分辨率遗传图谱的获取。在这项研究中,使用科农9204和京411为亲本的188个F8:9代重组自交家系(recombinant inbred line, RIL)构建了高密度遗传图谱(图1)。得到的遗传图谱由119,566个位点组成,总遗传距离为4424.4cM。其中119,001个为SNP标记,来自于小麦660K SNP基因分型芯片。该图谱与国际小麦基因组测序协会发表的中国春全基因组组装序列及利用小麦90K和小麦820K SNP芯片构建的整个图谱共线性关系良好。高密度小麦遗传图谱为小麦遗传和基因组研究提供了重要的资源。
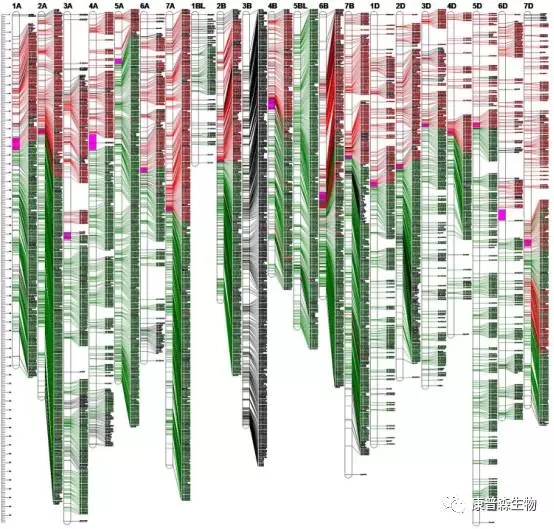
图1 小麦高密度遗传连锁图
同时,对构图SNP在基因组上的位置进行了分析。图2圈图中左上表示SNP位点在每条染色体上的比例;右上表示着丝粒附近SNP位点的比例;左下表示编码区与非编码区的分布比例;右下表示每条染色体遗传图谱的遗传距离。图3对构图SNP位点的遗传位置与物理位置进行了一一比对。
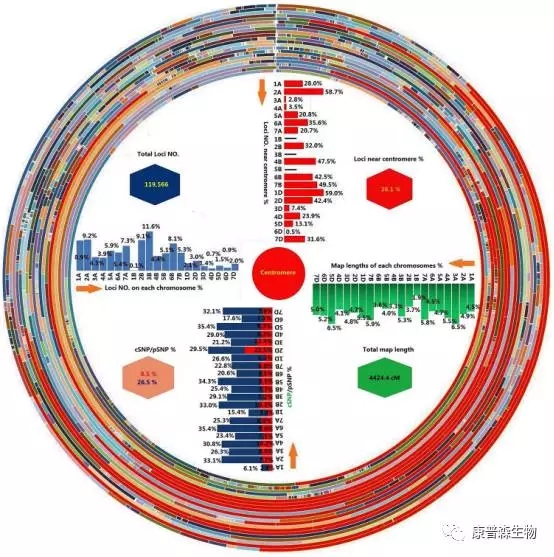
图2 构图SNP位点在基因组上的分布情况
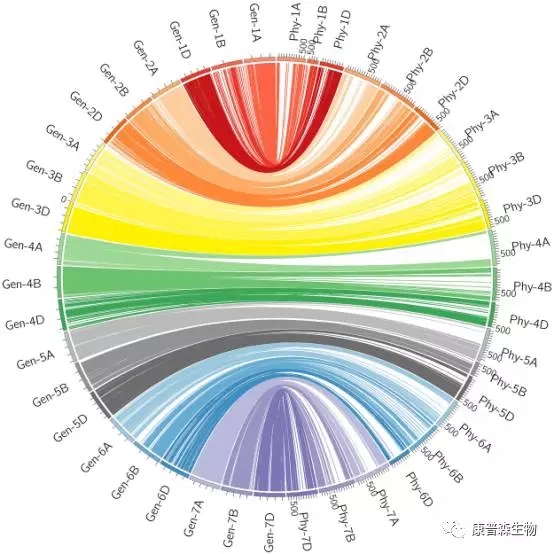
图3 构图SNP遗传与物理位置对比图
此外,该研究对产量相关性状的——穗粒数的主效QTL基因进行了定位。使用MapQTL 6.0, IciMapping 4.1, and QTLNetwork 2.0等软件对群体在10个不同环境下的穗粒数表型数据进行分析,qknps-4a基因被定位在4A染色体上,解释表型变异率为8–21.2%。不同分析软件分析的重叠置信区间为149.6–150.3 cM,标记Ax-109844107和Ax-110540586之间图4,对应物理位置为4:680398739–4:683638403,此区段内预测有65个基因。经分析亲本京411携带的等位基因可增加穗粒数1.2–2.4。该研究为今后目标QTL的精细定位及图位克隆奠定了基础。
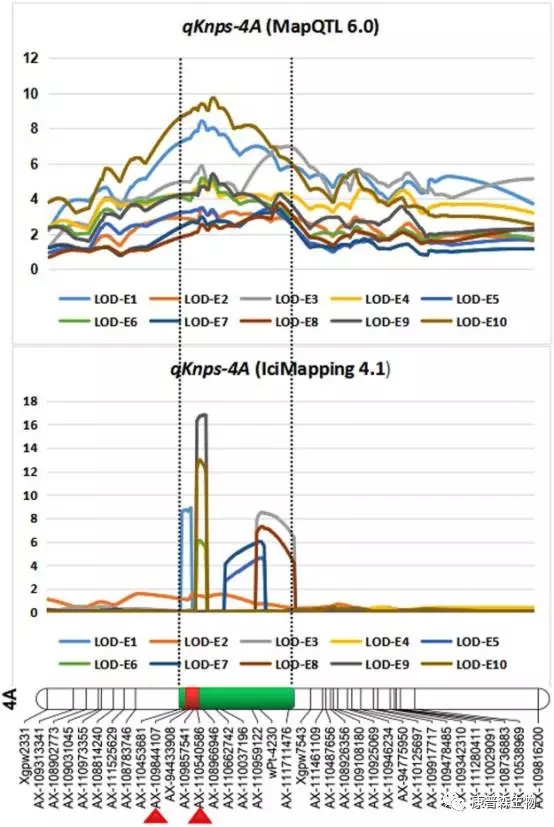
图4 不同环境下qknps-4a基因定位的置信区间
原文链接:
Cui F, Zhang N, Fan X, et al. Utilization of a Wheat660K SNP array-derived high-density genetic map for high-resolution mapping of a major QTL for kernel number[J]. Scientific Reports, 2017, 7(1):3788.
https://www.nature.com/articles/s41598-017-04028-6.pdf

 PREV
PREV
